>1100万条序列!INDI—迄今最全纳米抗体数据库,网站全公开,数据可离线下载
纳米抗体的小尺寸赋予了多种治疗优势(稳定性、肿瘤渗透性),这些小爱已在前文介绍多次,2018 年纳米抗体首次获得治疗批准,巩固了这种形式的临床可行性。纳米抗体的结构化数据和序列信息将使基于纳米抗体的疗法的临床开发加速,尽管纳米抗体序列和结构数据正在被加速公开,但来源的异质性和缺乏标准化阻碍了纳米抗体信息的可靠收集。
为了有效整合当前已公开的纳米抗体的庞大数据库,近日,来自美国和波兰的开发者创建了用于免疫信息学的纳米抗体集成数据库(INDI,http://naturalantibody.com/nanobodies),INDI基于专利、GenBank、NGS库以及结构科学出版物四大主要序列渠道搜集纳米抗体序列信息,并配备强大的纳米抗体特定序列和文本搜索,可访问超过 1100 万纳米抗体序列,有助于实现基于纳米抗体形式的药物的治疗前景。
数据采集
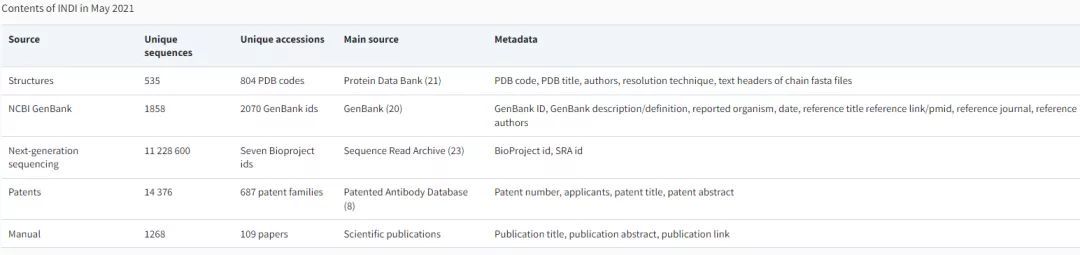
INDI中的数据来源
INDI开发者们绘制了最常见的检索任务,以促进与 INDI 在线和离线的交互。登录网址http://naturalantibody.com/nanobodies,用户可以执行基于纳米抗体特定序列的搜索和元数据检索。为了便于离线免疫信息学分析,开发者还提供大量的数据下载。
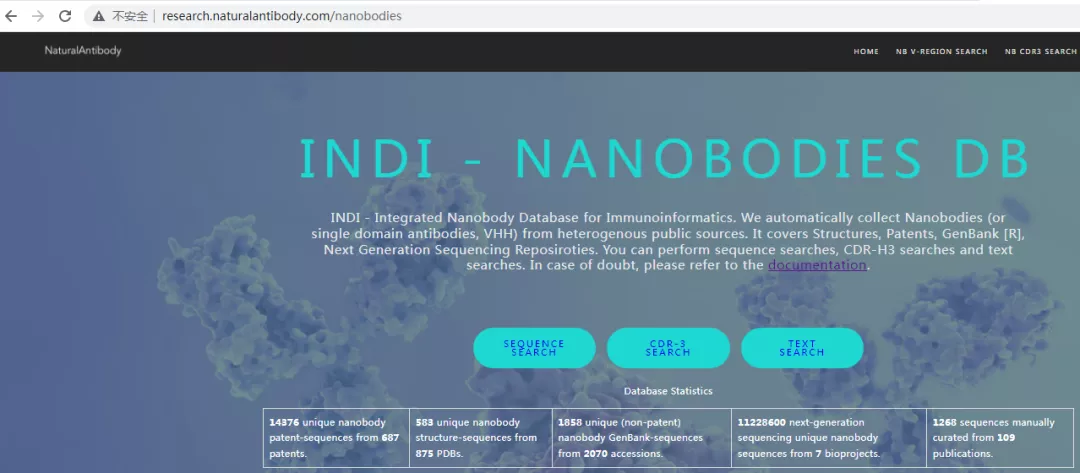
基于序列的搜索
INDI提供了两个纳米抗体特定的序列搜索功能,以促进与 INDI 中数据的交互——可变区域搜索和CDRH3搜索。这种划分反映了纳米抗体序列识别的两个常见用例。前者解决了整个可变区的检索,后者针对的是产生大多数抗原接触的纳米抗体中最可变的区域,即CDRH3的具体搜索。
可变区域搜索用于检索与查询最匹配的整个纳米抗体序列。为了反映搜索的纳米抗体特异性,开发者使用 IMGT 方案比较纳米抗体序列,该方案为抗体/纳米抗体的比对提供了免疫球蛋白特异性框架。查询序列是 IMGT 编号的,随后根据 IMGT 位置与 INDI 中预先编号的纳米抗体序列对齐。结果按整个可变区的最高序列同一性排序并以交互式可排序表展现,该表可在每次点击时获得更详细的结果。
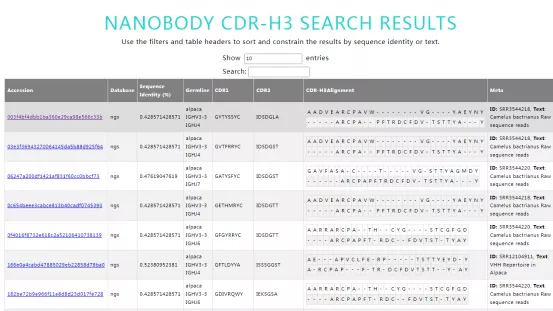
CDR3检索示例
考虑到在三个重链 CDR 中,CDRH3 位于抗体的抗原结合部位中心,开发团队为 INDI 配备了检索 CDRH3 不考虑其余可变区域的搜索工具。CDRH3 搜索的输入是 IMGT 定义的 CDRH3 序列,随后借助分子生物信息算法工具Biopython对结果进行比对。这使得类似cdr3s匹配的序列可以独立于长度检索。CDRH3结果显示在一个交互式的可排序表中,该表允许用户浏览结果并跟踪到可变序列及其相关元数据的链接。
文本搜索
INDI 中的纳米抗体序列与丰富的文本注释相关联,揭示了生物靶点、分子研究的起源和目的。元数据字段在源之间及其内部是异构的。例如,与结构相关的元数据将包含其他数据库中不存在的特定晶体参数。在 GenBank 中,关于纳米抗体靶点的信息可以包含在特定accession号或个别翻译的描述中,这是因为没有标准方法来报告此类信息。早期捕获抗体/纳米抗体靶点信息的努力依赖于大规模的手动管理。鉴于 INDI 包含三个自动组件,对所有条目进行常规的手工注释是很有挑战性的。文本表示的多样性给文档检索带来了挑战。
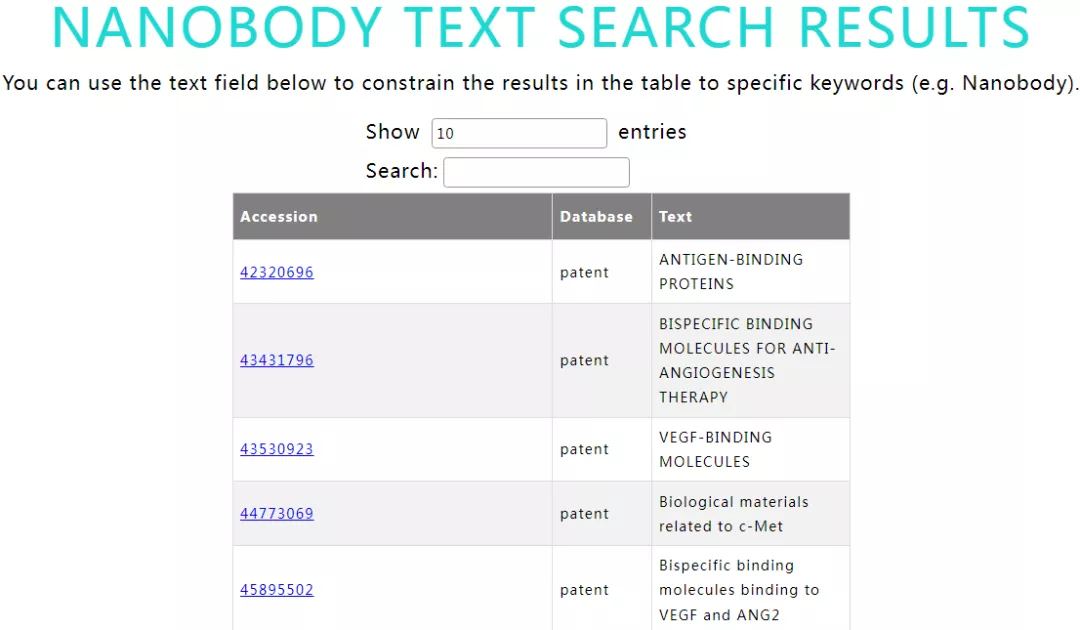
为了解决跨五个不同来源的信息检索问题,研究者实现了在所有数据库中的所有元数据字段上创建文本索引。用户仅需输入感兴趣的关键词,INDI 将检索与结果最匹配的条目,检索结果显示为一个交互式表,其中列出了条目、来源数据库和文本字段。用户可以对结果进行排序并显示匹配文本条目的详细信息。后者与链接到的纳米抗体序列accession号一起显示。
为了补充基于网络的检索,INDI开发者还提供数据供离线使用。序列提取物包含开发者鉴定的纳米抗体 V 区序列。每个序列条目都链接到元数据提取中包含的元数据字段。元数据字段也按五个数据库之一排序。所有数据均可通过 INDI 网站(http://naturalantibody.com/nanobodies)获得。
讨论
将抗体药物用于临床需要投入大量时间和资源,在临床试验阶段失败的可能性很高。具有良好生物物理特性的纳米抗体等新型形式为降低某些药物发现风险提供了机会。目前,靶向递送基于纳米抗体的疗法的创新方法正在被积极探索。除了分子疗法,纳米抗体还被用于开发多种细胞疗法。总而言之,使用传统基于实验室的方法开发纳米抗体疗法在被普及之前仍然需要进行多年的实验。而计算方法可以加速这一过程,更快地提供挽救生命的疗法。
尽管仍处于起步阶段,但科研工作者正在开发解决治疗性纳米抗体设计问题的生物信息学方法。计算纳米抗体方法可以为开发可靠的结构建模方法、噬菌体展示库的设计或新型纳米抗体的计算设计提供见解。基于此,开发者创建了INDI网站,这是一个将纳米抗体序列、结构及其相关元数据整合到公共领域的数据库。来自异构源的自动更新可以跟上公共领域的沉积步伐。INDI 中数据的异质性使纳米抗体研究人员能够准确了解纳米抗体序列、结构和功能的当前状态,可以加速分析框架、结构建模以及从头纳米抗体药物设计的开发,并为解决纳米抗体设计的深度学习模型提供基础。总之正如开发者期待的,希望 INDI 将形成坚实的数据基础,以开发纳米抗体特定的计算方法,从而加速基于纳米抗体的新型疗法的开发。

